迅速簡便ウイルス検出・ウイルス汚染箇所可視化ツール – PULSERAA – を開発
迅速簡便ウイルス検出・ウイルス汚染箇所
可視化ツール – PULSERAA – を開発
国立大学法人東京農工大学、東海大学、ノースカロライナ大学、フマキラー株式会社研究開発本部の研究者らによる国際共同研究チームは、ウイルスを迅速簡便に検出し、さらに検出試薬を噴霧するのみでウイルス汚染箇所を可視化する基盤技術 (Proximity-Unlocked Luminescence by Sequential Enzymatic Reactions from Antibody and Antibody/Aptamer: PULSERAA) を開発しました。実際に不活化SARS-CoV-2や不活化インフルエンザウイルスを検出した結果、試薬を混合するのみでそれぞれ高感度な検出を達成しました。また、ニトロセルロース膜に滴下した不活化SARS-CoV-2に対して試薬を噴霧し、スマートフォンのカメラで撮影したところ、ウイルス量依存的な化学発光を確認することができました。試薬を混合または噴霧するのみでウイルス汚染箇所が可視化されることから、誰でも簡単・確実な感染制御が可能であり、今後のパンデミックなどでの感染拡大防止や感染予防に貢献すると期待されます。
本研究成果は、Advanced Science(9月24日付)にオンライン掲載されました。
論文タイトル:Proximity-Unlocked Luminescence by Sequential Enzymatic Reactions from Antibody and Antibody/Aptamer (PULSERAA): A Platform for Detection and Visualization of Virus-Containing Spots
URL:https://doi.org/10.1002/advs.202403871
背景
新型コロナウイルス (SARS-CoV-2) の世界的蔓延によって、日々の感染制御の在り方が大きく変わりました。また、人と人との身体的な接触を余儀なくされる病院や老人介護施設、保育所や幼稚園などにおいてクラスターが発生したことで、身体的被害のみならず風評被害などによる経済的・精神的被害も多く見られました。このようなクラスターは、患者からウイルスがどのように飛散し、どの場所にどの程度存在するかを即時に判断できないことで生じます。
そこで本研究では、ウイルスの迅速簡便な検出のみならず、試薬を噴霧し、スマートフォンに内蔵されているカメラを通して撮影することで、ウイルスが含まれる箇所、即ちウイルスに汚染された箇所を可視化することができるような検出方法の確立を目指しました。
研究体制
本研究は、東京農工大学大学院グローバルイノベーション研究院の三浦大明特任助教、東京農工大学大学院工学研究院生命機能化学部門の浅野竜太郎教授、塚越かおり助教、Vavricka J. Christopher准教授、津川若子准教授、池袋一典卓越教授、工学府生命工学専攻大学院生 (当時) の林和佳奈、佐々木一慧、工学府産業技術専攻大学院生 (当時) の平野健輔、東京農工大学農学部附属感染症未来疫学研究センターの水谷哲也教授、竹前等准教授、東海大学医学部基盤診療学系臨床検査学の浅井さとみ教授、柿添英文非常勤医師、ノースカロライナ大学チャペルヒル校の早出広司卓越教授、およびフマキラー株式会社研究開発本部との共同研究によって行われました。
本研究は科学技術振興機構 (JST) 未来社会創造事業探索加速型研究費JPMJMI19D2、日本学術振興会 (JSPS) 科研費21K18321、新エネルギー・産業技術総合開発機構 (NEDO) 研究費JPNP20004、フマキラー株式会社開発本部の支援を受けて実施されました。
研究成果
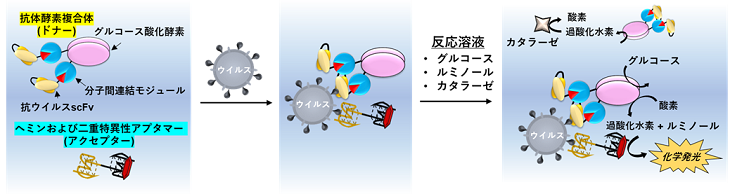
抗体を分子認識素子とした一般的な検出系では、抗原特異的なシグナルを得るために、抗原と結合していない分子認識素子を洗浄操作などによって除去する必要がありますが、操作が煩雑かつ複雑であるため、しばしば熟練した技術者によって行われる必要があります。誰もが簡単に検出することができるようになるためには、このような煩雑な実験操作を必要としない検出法の設計が必要です。本研究では、ウイルス表面上というごく限られた場所でのみ効率的に2分子(ドナーとアクセプター)間で連続的な酵素反応が発生し、化学発光が観察できるような分子設計を行うことにより、ドナーとアクセプターがウイルス表面上で近接した時のみ化学発光を生じ、煩雑な操作を必要としない検出系Proximity-Unlocked Luminescence by Sequential Enzymatic Reactions from Antibody and Antibody/Aptamer (PULSERAA) を構築しました (図1)。ドナーとして、ウイルス表面抗原結合低分子抗体とグルコース酸化酵素を分子間連結モジュール(1)によって連結した抗体酵素複合体を用いました。グルコース酸化酵素の酵素反応によって、グルコースを基質として過酸化水素が発生します。アクセプターとしては、過酸化水素とルミノールを基質として化学発光を生じる反応を触媒するペルオキシダーゼ活性を持つ分子を用いました。この分子の一つとして用いたヘミンは、分子認識能を持つDNAであるアプタマーと結合することで、そのペルオキシダーゼ活性が大幅に向上することが明らかとなっています(2)。そのため、ヘミンとウイルスに結合する二重特異性アプタマーを開発し、アクセプターとして用いました。
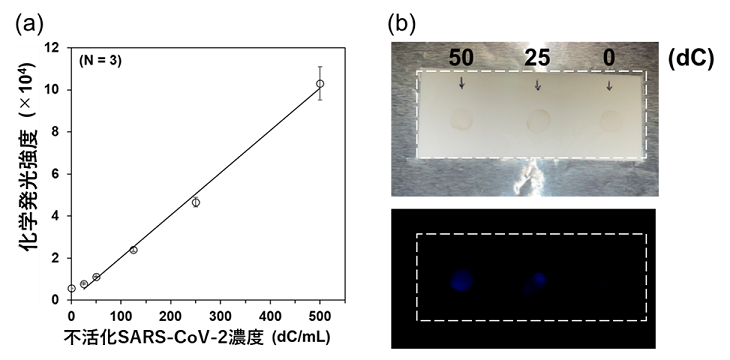
ドナーとアクセプター、および不活化SARS-CoV-2を混合した後、それぞれの基質となるグルコース、ルミノール、およびカタラーゼ(ウイルスに結合していない抗体酵素複合体から産生される過酸化水素を除去し、バックグラウンドシグナルを低減するため)を含む反応溶液を添加し、プレートリーダーで化学発光を定量しました。その結果、操作時間約15分で25-500 デジタルコピー (dC)/mLの濃度範囲で高い直線性が確認されました (図2a)。これはこれまでSARS-CoV-2の確定診断で用いられてきた定量PCRによる検出感度とほぼ同等の感度でした。また、各種条件検討を行った結果、ニトロセルロース膜上に固定した不活化SARS-CoV-2の量依存的な化学発光を、スマートフォンのカメラを通して撮影することに成功しました (図2b)。
本検出系は、ドナーである抗体酵素複合体に分子間連結モジュールを、アクセプターには化学合成が容易な二重特異性アプタマーを用いていることから、それぞれ異なるウイルスに対する低分子抗体とアプタマーに変更することが可能です。実際に、幅広いSARS-CoV-2変異体に結合することが可能な低分子抗体に変更することで、不活化SARS-CoV-2オミクロン株や、感染患者から採取した鼻拭い液中に含まれるベータ株の検出にも成功しました。さらに、それぞれをA型インフルエンザウイルス表面抗原に結合するものに変更することで、不活化インフルエンザウイルスも同様に検出できることが確認されました。
今後の展開
本研究では、試薬を混合するのみでウイルスを迅速かつ高感度で検出し、さらに試薬を噴霧することでウイルスが付着している箇所を、デジタルカメラを通して観察することで同定可能な検出系PULSERAAを構築しました。操作時間が約15分と現状のイムノクロマトグラフィーと同程度の時間で検出が可能ですが、噴霧の場合はさらなる時間短縮が求められることから、より強力な分子認識素子の開発などが必要であると考えています。PULSERAAは分子認識素子を簡便に他のウイルスに対するものに変更することができる設計であるため、今後起きうるパンデミックに対する感染制御プラットフォームとなることが期待されます。
(1) Zakeri et al., Proc. Natl. Acad. Sci., 2012, 109, E690-E697.
(2) Travascio et al., Chem. Biol., 1998, 5, 505-517.
◆研究に関する問い合わせ◆
東京農工大学 大学院工学研究院
生命機能科学部門 教授
池袋 一典(いけぶくろ かずのり)
TEL/FAX:042-388-7030
E-mail:ikebu(ここに@を入れてください)cc.tuat.ac.jp
関連リンク(別ウィンドウで開きます)
